Liens utiles:
1. TECHNIQUE D'ELECTROPHORESE
- 2. MARQUEURS BIOCHIMIQUES ET MOLECULAIRES
La phylogénie moléculaire
est basée sur l'analyse comparative des séquences
d'ADN ou de protéines. Elle compléte les méthodes
traditionnelles de classification basées sur l'observation des
caractères morphologiques et anatomiques.
La phylogénie moléculaire utilise les gènes
des organismes vivants pour élaborer des arbres
phylogénétiques en comparant les caractères
moléculaires (séquences nucléotidiques ou protéiques).
La phylogénie moléculaire essaie de retracer l'accumulation
des mutations dans les génomes au cours de l'évolution
des espèces. Elle permet de reconstruire l'histoire évolutive
des organismes. Les espèces ont des génomes d'autant plus
proches qu'elles ont divergé récemment depuis leur ancêtre
commun.
Important: Pour comprendre ce cours, il faut réviser la partie des techniques ELECTROPHORESE et MARQUEURS BIOCHIMIQUES ET MOLECULAIRES
NOYAU :
Génome nucléaire = Génome
PLASTES : Génome plastidial = Plastome
MITOCHONDRIES:
Génome mitochondrial = Mitochondriome
Transcriptome:
ensemble des ARN (messagers, ribosomiques, de transfert et autres espèces
d'ARN) issus de la transcription du génome.
Les
régions du génome utilisées pour la phylogénie
moléculaire sont appelées marqueurs moléculaires.
Elles doivent remplir les conditions suivantes:
1/ elles doivent être ubiquistes, c'est-à- dire présentes
chez tous les taxons étudiés
2/ elles doivent contenir une région variable spécifique
de chaque espèce et encadrée par deux séquences
conservées.

La
comparaison des séquences des régions variables des différentes
espèces permet d'apprécier leur proximité ou leur éloignement phylogénétique.
La phylogénie moléculaire est de plus en plus utilisée
pour l'identification et la classification des êtres vivants.
En
plus des gènes codant pour des protéines, Les gènes donnant des ARNs sont très importants pour la phylogénie moléculaire.
ARN ribosomique: L'ARN ribosomique est lui-même
produit à partir de gènes codés
dans l'ADN. Il représente 80 % de l'ARN total d'une cellule
(ARN ribosomique, ARN messager, ARN de transfert). Les ARN ribosomiques
sont transcrits sous forme de précurseurs plus longs qui sont
clivés ensuite pour donner les différents ARNr. Chez les
eucaryotes, ce processus de maturation/clivage se déroule dans
le nucléole. Associé à des protéines,
les ARNr forment les ribosomes (dans le cytoplasme) qui permettent
la lecture de l'information génétique transcrite par l'ARN
messager. Au niveau des ribosomes, les ARNr sont repliés sur
eux-mêmes, formant une structure tridimensionnelle compacte qui
leu confère plus de protection. Ils deviennent ainsi
très stables par comparaison au ARN messagers.
L'ARN de la petite sous-unité du ribosome est une molécule
dont la séquence est très utilisée pour faire des
études de phylogénie moléculaire. On la trouve
conservée dans l'ensemble des organismes vivants. En comparant
les séquences du gène de cet ARN
(au niveau de l'ADN) chez différentes espèces, il devient
possible d'évaluer leur parenté évolutive. La base
de données du 'Ribosomal database project' (rdp.cme.msu.edu)
recense les séquences de l'ARN ribosomique 16S
(chez les Procaryotes) ou 18S
(chez les Eucaryotes) de plus de 270 000
espèces vivantes. Ces données permettent de reconstituer
un arbre phylogénétique des espèces.
L'ARNr 16S (constituant la petite sous-unité des ribosomes des procaryotes) s'est imposé comme marqueur de référence chez les procaryotes. Les gènes codant cet ARN (ARNr 16S) sont appelés ADNr 16S (16S rDNA en anglais) et leur séquence est très utilisée en phylogénie.
Le plastome est souvent utilisé dans le séquençage
et la phylogénie moléculaire des plantes, car:
- Il a une taille réduite
- Il existe sous forme de milliers de copies par cellule.
- Il est hautement conservé
La transmission du génome plastidial d'une génération
à la suivante est non mendélienne (transmission
cytoplasmique). Elle est essentiellement maternelle.
Une cellule végétale possède en moyenne une centaine
de chloroplastes et un chloroplaste possède une centaine de copie
d'ADNcp (molécule circulaire). Ainsi,
une cellule possède environ 10 000 copies d'un gène chloroplastique.
La taille moyenne de l'ADNcp est comprise entre 120 et 160 kpb.
L'ADN
mitochondrial (ADNmt). La transmission de l'ADNmt est généralement
non mendélienne. Dans la plupart des cas, il est transmis
par la mère. Cependant il existe de nombreuses exceptions
chez les plantes, les champignons et même chez les animaux.
Les gènes mitochondriaux, et en particulier le gène codant
pour la sous-unité I de la cytochrome c oxydase (CoxI),
sont utilisés chez les eucaryotes.
De même, le gène du cytochrome b
porté par le mitochondriome a été utilisé
pour comparer plusieurs espèces.


Le transcriptome est l'ensemble des ARN (messagers, ribosomiques, de transfert et autres espèces d'ARN) issus de la transcription du génome. L'analyse transcriptomique peut caractériser le transcriptome d'un tissu particulier, d'un type cellulaire, ou comparer les transcriptomes entre différentes conditions expérimentales.
Par opposition au ARNr, les ARN messagers de structure fragile, ont une durée de vie courte.
Séquençage de l'ADN selon la méthode de Sanger
Le
le séquençage de l'ADN est la détermination de la succession des nucléotides le composant.. Cette technique exploite les connaissances acquises depuis
une trentaine d'années sur les mécanismes de la réplication de l'ADN. Une amorce marquée à la
radioactivité P32 est utilisée dans la polymérisation.
Les réactions de polymérisation pour synthétiser un
ADN complémentaire à l'ADN à séquencer se font par addition de désoxyribonucléotides (dNTP) et des nucléotides légèrement différents : les didésoxyribonucléotides (ddNTP).
Les ddNTP diffèrent des dNTP par l'absence d'un groupement OH
bien précis. En effet, lorsqu'une ADN polymérase utilise
un ddNTP au lieu d'un dNTP, elle n'est plus capable de rajouter le moindre
nucléotide à sa suite : la synthèse du brin d'ADN
s'arrête.

A
un moment aléatoire, un ddNTP sera ajouté à la
chaîne en cours de synthèse par l'ADN polymérase.
Cette synthèse s'arrêtera à cet endroit.
On réalise la polymérisation dans 4 tubes correspondant chacun
à un ddNTP différent.
Par exemple, si le milieu réactionnel contient une faible proportion
de didésoxyribonucléotide à Guanine (ddGTP), on
obtiendra à la fin des réactions un ensemble de brins
d'ADN de tailles variés, selon l'endroit où un ddGTP se
sera inséré et que la réaction aura ainsi été
stoppée (ce qui correpond, du fait de la complémentarité
des bases, à la présence d'une Cytosine dans le brin d'ADN
séquencé). On répète la même opération
avec un milieu contenant du ddATP, un milieu contenant du ddCTP, et
un milieu contenant du ddTTP.
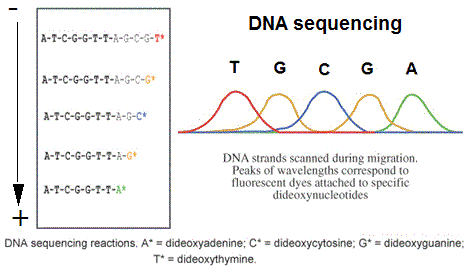
Au
lieu du marquage radioactif, chaque ddNTP (ddATP,ddTTP,ddCTP,ddGTP)
est marqué par un fluorochrome différent dont le
spectre d'émission est spécifique. Une analyse spectrale
va différencier les différents fluorochromes, associer
la base correspondante et donc définir la séquence nucléotidique
du brin d'ADN initial. Les brins allongés se terminent par
un didéoxynucléotide marqué par un fluorochrome
différent selon la base présente (A,T,C, G ).
La taille des fragments obtenus est déterminée par une
chromatographie d'exclusion au lieu de l'électrophorèse.
Le séquenceur détecte la florescence sortant des colonnes
de chromatographie (4 colonnes), repérant les fragments d'ADN
et leurs tailles précises. Les systèmes les plus modernes
permettent même de lire les quatres nucléotides à
partir d'une seule colonne de chromatographie.




Automatisation du séquençage (suite)
La
chromatographie d'exclusion
va permettre le piegeage de particules de faible poids
moléculaire sur une colonne Sephadex G50
constituée de billes perforées dont les trous ont un diametre
determine (ici de 20 a 50 µm). Les petites particules de diamètre
inferieur à ceux-ci entrent et sont piégées. A
l'inverse, les molécules de grande taille vont passer autour
et seront éluées très rapidement.
Les produits de la reaction de polymérisation sont purifiés
par chromatographie pour pieger les ddNTP libres (taille faible), en
excès. En effet ces ddNTP libres non incorpores lors de la reaction
pourraient parasiter les signaux de fluorescence specifiques. De même,
les sels eventuellement présents sont piégés de
la même manière que les ddNTP. Le
résultat est présenté par la machine sous forme
de courbes montrant la fluorescence détectée (et l'équivalent
en terme de nucléotides).
Les séquences écrites en ligne sont envoyées sur Internet et comparées à des séquences déposées dans des banques de données. Des BLAST sont ainsi obtenus, c'est à-dire des comparaisons avec les séquences les plus proches.

Ainsi, pour le cas des ARNr, la banque RDP (Ribosomal Database Project II) a été développée afin de répondre à la demande croissante d'analyse à haut débit des séquences d'ARNr.
Phylogénie moléculaire par PCR-RFLP entre Homme, Chimpanzé, Gorille et Orang-Outan. Exemple introductif
Au lieu du séquençage, on utilise une PCR-RFLP. Le fragment d'ADN étudié est dans un premier temps amplifié par PCR, avant d'être soumis à l'hydrolyse par une enzyme de restriction.
Exemple d'étude
Les gènes BRCA-1 et 2 (pour "BReast CAncer" ou cancer du sein) sont présents chez tous les mammifères et sont responsables de la régulation des cycles de division cellulaire. Les gènes BRCA-1 et BRCA-2, situés sur les chromosomes 17 et 13, respectivement, inhibent la division cellulaire anarchique. Ils jouent le rôle de gènes suppresseurs de tumeurs. Certaines mutations de ces gènes peuvent accroître le risque de développement d'un cancer du sein.Etude chez les primates:

Technique moléculaire: RFLP, une seule enzyme de restriction

Analyse RFLP Gène BRCA-1 sur gel d'agarose (E-gel 1,2% d'agarose)
Interprétation des résultats:
-
Les profils de restriction RFLP ont été obtenus au moyen
d'une seule enzyme de restriction.
- Les profils de l'homme et du chimpanzé
sont identiques.
- Un seul fragment est commun entre les profils
de l'Homme, du Chimpanzé et de celui du Gorille.
- Le profil de l'orang-outan est unique et ne présente aucun fragment en commun avec les trois autres espèces.
Conclusions:
- L'Homme partage un ancêtre commun plus proche avec le Chimpanzé qu'avec le Gorille et l'Orang-outan.
- Les Hominidés constitué de l'Homme et du Chimpanzé partage un ancêtre commun plus proche avec le Gorille que l'Orang-outan.

Ouvrages de la préparation de la transition Secondaire-Supérieur


Video 'genetic diversity through molecular markers' (master level, Cadi Ayyad University, 2019:
-
من علوم الحياة إلى البيوكيمياء
Des Sciences de la vie au Lycée à la Biochimie à l'Université
-
تحميل ملفات في مواضيع البيوكيمياء
Téléchargement de fichiers en Biochimie
BIOCHIMIE. TP CONSIGNES (Vidéos)
Chaine Youtube (abonnement). Plusieurs vidéos multilingues

ANNONCES, INFORMATIONS
Congrès, Conférences,...
- Congrès, Conférences
- Cours, Workshops
- Bourses, Postdocs
- Biotech-ecolo annonces
CONCOURS:
Concours de Pharmacie
Afin de pouvoir continuer à servir les visiteurs, soutenez nos actions sur le site takween en faisant acquisition des ouvrages et supports pédagogiques desitinés à améliorer l'enseignement et la recherche scientfique en Biochimie.